Iodothyronine deiodinase 1 (DI1)
La DI1 és una selenoproteïna que es troba anotada a selenoDB en Myotis lucifugus, i al ser aquesta espècie del mateix gènere que la nostra espècie d'estudi (Myotis davidii), deduïm que és la més apropiada per a ser comparada.
El blast ens dona tres hits diferents, el primer i el segon en el mateix sentit a l'anotació (+) i el tercer en el mateix sentit a l'anotació (-). D'aquests tres hits, preveiem que la nostra selenoproteïna es troba dins de l'scaffold KB110890.1, ja que presenta la més alta homologia amb la query i una puntuació de tcoffe molt elevada (score 100).
Tant l'exonerate com el genewise han predit tots dos una proteina. En el cas de l'exonerate té 4 exons i 3 introns, amb una raw score de 1262. El genewise, prediu també una proteïna de 4 exons i 3 introns, amb un score de 534.62 bits.
L'estructura predita mitjançant exonerate té un codó stop a la mateixa posició que la selenoproteïna de la query (aminoàcid 121). Pel que fa al tcoffee, com ja hem dit, trobem una puntuació alta que ens reafirma que la estructura està ben conservada.
Pel que fa a la predicció d'elements SECIS, el SECISearch3 ens prediu un element SECIS a la posició 3', que ens valida la teoria que DI1 és una selenoproteïna en Myotis davidii. D'altra banda, SEBLASTIAN ens confirma que en el nostre subseq trobem DI1, mostrant l'homologia del nostre scaffold amb la DI1 de Ovis aries. Amb tota aquesta informació, podem predir l'estructura de la selenoproteïna:

Iodothyronine deiodinase 2 (DI2)
La DI2 és una selenoproteïna que es troba anotada a selenoDB en Myotis lucifugus, i com aquesta espècie i la nostra espècie d'estudi (Myotis davidii) són del mateix gènere és la més apropiada per comparar-la amb la nostra.
El blast ens dona tres hits diferents, el primer en sentit contrari a l'anotació (-) i el segon i el tercer en el mateix sentit a l'anotació (+). D'aquests tres hits, preveiem que la nostra selenoproteïna es troba dins de l'scaffold KB199564.1, ja que presenta la més alta homologia amb la query i una puntuació de tcoffe molt elevada (score 100).
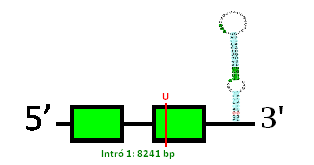
Tant l'exonerate com el genewise han predit tots dos una proteina. En el cas de l'exonerate té 2 exons i 1 intró, amb una raw score de 1365. El genewise, prediu també una proteïna de 2 exons i 1 intró, amb un score de 614.42 bits.
L'estructura predita mitjançant exonerate té un codó stop a la mateixa posició que la selenoproteïna de la query (aminoàcid 134). Pel que fa al tcoffee, com ja hem dit, trobem una puntuació alta que ens reafirma que la estructura està ben conservada.
Pel que fa a la predicció d'elements SECIS, el SECISearch 3 ens prediu un element SECIS a la posició 3', que ens valida la teoria que DI2 és una selenoproteïna en Myotis davidii. D'altra banda, el SEBLASTIAN no ens ha proporcionat cap resultat, però com que s'ha trobat estructura SECIS i selenocisteïna en la seqïència gènica, deduïm que Seblastian ha comés un error de predicció i que DI2 és una selenoproteïna.Amb tota la informació obtinguda hem predit l'estructura de la selenoproteïna:
Iodothyronine deiodinase 3 (DI3)
La DI3 és una selenoproteïna que no es troba anotada a selenoDB en Myotis lucifugus. és per això, que hem utilitzat la query de la selenoproteïna de Homo sapiens.
El blast ens dona tres hits diferents, el primer i tercer en sentit contrari a l'anotació (-) i el segon en el mateix sentit a l'anotació (+). D'aquests tres hits, preveiem que la nostra selenoproteïna es troba dins de l'scaffold KB109558.1, ja que presenta una alta homologia amb la query humana i una puntuació de tcoffe molt elevada (score 99).
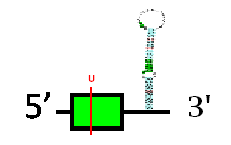
Tant l'exonerate com el genewise han predit tots dos una proteïna. En el cas de l'exonerate té 1 sol exó i cap intró, amb una raw score de 1113. El genewise, prediu també una proteïna de 1 sol exó i cap intró, amb un score de 504.80 bits.
L'estructura predita mitjançant exonerate té un codó stop a la mateixa posició que la selenoproteïna de la query (aminoàcid 144). Pel que fa al tcoffee, com ja hem dit, trobem una puntuació alta que ens reafirma que la estructura està ben conservada.
Pel que fa a la predicció d'elements SECIS, el SECISearch 3 ens prediu un element SECIS a la posició 3', que ens valida la teoria que DI3 és una selenoproteïna en Myotis davidii. D'altra banda, SEBLASTIAN ens confirma que en el nostre subseq trobem DI3, mostrant l'homologia del nostre scaffold amb la DI3 de Homo sapiens. Amb tota aquesta informació, podem predir l'estructura de la selenoproteïna:
Cal remarcar que la DI3 no es troba anotada en Myotis lucifugus (no es troba a SelenoDB), però si que es troba en la nostra espècie d'estudi, Myotis davidii. Donat que en l'ancestre comú (Homo sapiens) si que la trobem, hipotetitzem que es tracta d'una estructura que Myotis lucifugus ha perdut posteriorment a la diferenciació de aquest i la nostra espècie
